| PCRプライマーを設計する | |||||||
| Title | PCRプライマーを設計する | ||||||
| Caption | 実験対象となる生物種のゲノム配列で一ヶ所だけが増幅されるプライマー、つまりユニークなプライマーを設計する方法を説明します。 | ||||||
| Detail | 『PCRプライマーを設計する』 実験対象となる生物種のゲノム配列において一ヶ所だけが増幅されるプライマー、つまりユニークなプライマーを設計する方法を説明します。 【適切なプライマー】 PCRを用いれば、ゲノム配列上の特定領域を増幅することが可能です。ただ、適切なプライマーを用いなければ、狙った領域(以下、ターゲット領域)以外も増幅されてしまい、結局得られる増幅産物には様々な配列パターンが混在していることになります。 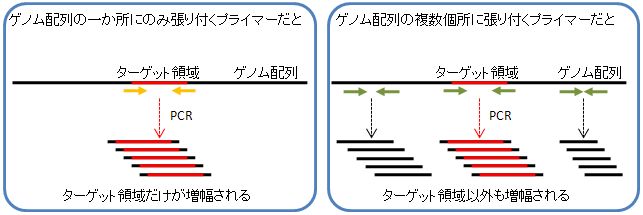 【ユニークであることの保証】 増幅したい領域のゲノム配列が得られれば、Primer3等のプライマー設計ツールを用いて、プライマー配列を求めることができます。ただ、このプライマー配列はターゲット領域だけを増幅するものであると保証されているわけではありません。そこで、Primer3等で得られたプライマー配列が、ゲノム配列上で一ヶ所にしか張りつかないことを確認します。この確認にはe-PCR等のプライマーマッピングツールを用います。 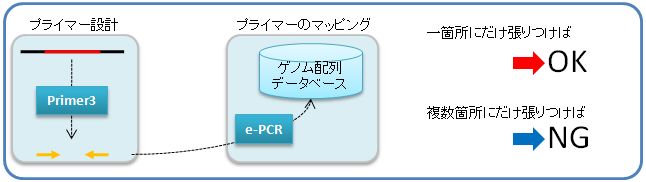 【WWWサイトの利用】 Primer3とe-PCRはインターネットを通じて利用可能です。以下に、それらのWWWサイトを利用してユニークなプライマーを設計する手順を説明します。
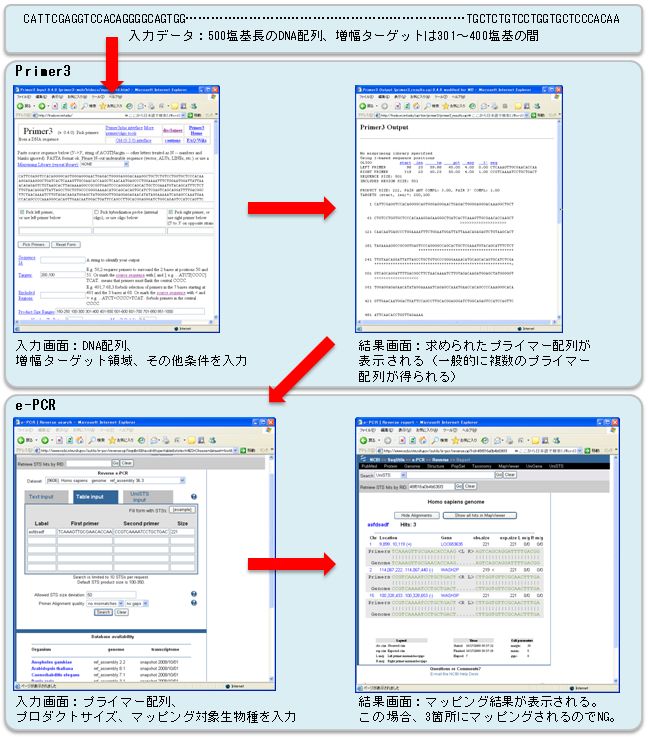 使用できるツール:
|
||||||